Une illustration du Deep Learning : Monitorer son activité quotidienne en temps réel – 2nde partie
Cette seconde partie du billet s'intéresse à la mise en œuvre à proprement parlé de la solution décrite dans la première partie sur la base du jeu de données issu de l'UCI.
Pour ce faire, et comme précédemment indiqué, nous vous proposons d'utiliser le projet CNTK (Computational Network ToolKit) développé par Microsoft Research et disponible sur le repo/la forge communautaire GitHub.

Ce choix est notamment motivé par le fait que CNTK ait notamment démontré son efficacité via la compétition ImageNet (vision par ordinateur) dont il est sorti vainqueur en décembre 2015. Il est donc un allié de choix dans l'optique de développer votre besoin autour des méthodes proposées par le domaine du Deep Learning.
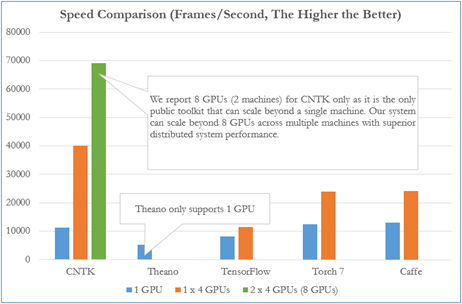
Figure 1 : Performance de CNTK vis-à-vis des autres bibliothèques disponibles
Il est temps de rentrer dans le vif du sujet et de poursuivre les présentations du protagoniste du moment, à savoir CNTK.
A propos de CNTK
CNTK est un projet Open Source notamment annoncé dans les billets Microsoft Releases Open Source Deep Learning Toolkit on GitHub et Microsoft releases CNTK, its open source deep learning toolkit, on GitHub.
Ce dernier permet de mettre en place facilement des algorithmes tirant profit de la puissance du Deep Learning comme précédemment développé dans le billet Une première introduction au Deep Learning.
Pour cela, ce projet exploite les différentes méthodes actuelles pour fournir le plus rapidement possible les résultats attendus. A ce propos, CNTK dispose de plusieurs points intéressants, notamment la possibilité d'utiliser de manière transparente, un CPU classique, mais aussi la puissance des GPU pour réaliser les calculs.
Il propose un langage de définition de haut niveau permettant de matérialiser les différentes couches et transformations qu'opérera le réseau de neurones. ( Les fichiers de définition du réseau sont fournis avec ce billet ici) .
Enfin, le Framework fournit un ensemble d'outils permettant de contrôler la manière dont l'apprentissage du modèle sera fait, notamment au travers du bloc SGD qui référence les paramètres influant sur la descente de gradient, et la manière dont seront générés les batchs d'entrainements.
Mise en place
Le projet CNTK est disponible sur le repo/la forge communautaire Github ici. A partir de là, deux possibilités s'offrent à vous :
- Récupérer les binaires précompilés et prêts à l'emploi
- Cloner le projet Git et le compiler par vous-même. Cela permet aussi de pouvoir contribuer au projet :-)
Dans tous les cas, vous obtiendrez un fichier exécutable nommé CNTK.exe qui sera notre point d'entrée.
C'est cet exécutable qui vous permettra d'entrainer vos modèles et de les interroger par la suite. En termes de mise en œuvre, nous vous conseillons d'ajouter le chemin vers le dossier parent de l'exécutable à votre variable d'environnement PATH.
Langage de définition de l'architecture de notre réseau de neurones
La véritable puissance de CNTK réside dans le langage évoqué plus haut et propre à ce dernier permettant de modéliser rapidement l'architecture des différentes couches et les opérations pour passer d'une couche à une autre dans notre réseau.
Les différentes caractéristiques du réseau et du processus d'entrainement sont exprimées au travers de macros dont nous allons détailler certaines d'entre elles par la suite.
Configuration globale de la solution
La première phase de la mise en place d'une solution via CNTK consiste à décrire le point d'entrée de celle-ci à l'instar d'un programme classique.
Dans ce contexte, certaines informations sont nécessaires afin de lui permettre d'appréhender l'environnement dans lequel il va s'exécuter.
Ces informations sont contenues dans un fichier .cntk, dont les premières lignes contiennent :
- La déclaration éventuelle des constantes.
- L'information de l'environnement d'exécution (CPU, GPU) : deviceId.
- Le plan d'exécution à suivre (l'enchainement des macros) : command.
- Les éventuels fichiers de définition des macros à charger : ndlMacros.
- Le niveau de trace souhaité : traceLevel.
Définition de la topologie du réseau de neurones
Modélisation simple
Une première approche simple permettant de mettre en œuvre un réseau de neurones simple consiste à expliciter uniquement le nombre de couches et le nombre de neurones par couche.
Cela est réalisé via la macro SimpleNetworkBuilder dont voici un exemple d'utilisation :
SimpleNetworkBuilder = [
# 2 input, 2 50-element hidden, 2 output
layerSizes = 2:50*2:2
trainingCriterion = "CrossEntropyWithSoftmax"
evalCriterion = "ErrorPrediction"
layerTypes = "Sigmoid"
initValueScale = 1.0
applyMeanVarNorm = true
uniformInit = true
needPrior = true
]
Script 1 : Exemple de définition d'un réseau de neurones
Dans l'illustration précédente, nous définissons les éléments suivants :
- layerSizes : cet élément précise le nombre de neurones pour chaque couche
- La syntaxe est la suivante : X : Y : Z où X Y Z représentent le nombre de neurones sur respectivement les couches 1, 2 et 3.
- Une syntaxe alternative (utilisée dans notre exemple) est : Y * n, laquelle permet de spécifier un réseau n couches avec le même nombre Y de neurones.
- Dans notre exemple : 2 : 50 * 2 : 2 est équivalent à 2 : 50 : 50 : 2
- trainingCriterion : Il s'agit de la mesure de performance qui sera utilisée à chaque itération
- evalCriterion : Il s'agit de la mesure de performance qui sera utilisée pour l'évaluation finale du modèle
- layerTypes : cet élément préciser la fonction d'activation à utiliser (Sigmoid ou Tanh)
- applyMeanVarNorm : cet élément indique s'il faut normaliser (centrer – réduire) les variables
Pour plus d'information sur chaque valeur, vous pouvez vous référer à la documentation afférente ici.
Vous l'aurez compris avec les explications précédentes, la macro SimpleNetworkBuilder nous permet de définir de manière simple la topologie ainsi que la manière dont sera évaluée la phase d'apprentissage.
Modélisation avancée
Si la macro précédente SimpleNetworkBuilder permet de modéliser rapidement des réseaux de neurones simples, il peut cependant s'avérer nécessaire dans des applications complexes de disposer d'un contrôle plus fin sur les différentes étapes et les connexions entre les couches.
C'est ce que propose justement la macro NDLNetworkBuilder. Cette dernière utilise pour cela un second fichier, dans lequel nous définissons la topologie du réseau ainsi que la manière dont sont connectées les couches. Il est ainsi possible de définir des opérations intermédiaires, telles que la convolution et/ou le pooling notamment utilisé dans les problématiques de reconnaissance d'images.
Vous avez ainsi la possibilité de définir dans un premier temps vos macros personnelles que vous pourrez ensuite réutiliser dans chacun de vos projets : ces macros pourront ensuite être importées et utilisées dans tous vos projets en utilisant le mot clé ndlMacros, facilitant ainsi l'extensibilité et la réutilisabilité de vos développements. Un exemple pratique peut être consulté ici.
Intéressons-nous maintenant à la façon dont seront enchainées les opérations entre nos différentes couches et à la manière d'expliciter tout cela avec CNTK.
Dans les faits, les opérations peuvent être représentées comme des listes chainées d'opérations, dont la sortie correspond à l'entrée de la suivante, etc. Par exemple, un réseau composé de 774 entrées, avec 3 couches cachées de 300 neurones puis une couche de sortie avec 7 valeurs possibles, peut être défini de la manière suivante :
ndlActivityTrackerMacros = [
nFeatures = 774
nLabels = 7
# Read features
features = Input(nFeatures, tag="feature")
# Normalize each feature (column-wise)
means = Mean(features)
invstd = InvstdDev(features)
featScaled = PerDimMeanVarNormalization(features, means, invstd)
# Read labels
labels = Input(nLabels, tag="label")
# Fully-connected layer with ReLU activation.
DnnReLULayer(inDim, outDim, x, parmScale) = [
W = Parameter(outDim, inDim, init = Gaussian, initValueScale = parmScale)
b = Parameter(outDim, init = fixedValue, value = parmScale)
t = Times(W, x)
z = Plus(t, b)
y = RectifiedLinear(z)
]
# Fully-connected layer with Sigmoid activation.
DnnSigmoidLayer(inDim, outDim, x, parmScale) = [
W = LearnableParameter(outDim, inDim, init="uniform", initValueScale=parmScale)
b = LearnableParameter(outDim, 1, init="uniform", initValueScale=parmScale)
t = Times(W, x)
z = Plus(t, b)
y = Sigmoid(z)
]
# Fully-connected layer with batch normalization and ReLU activation.
DnnBNReLULayer(inDim, outDim, x, wScale, bValue, scValue, bnTimeConst) = [
W = LearnableParameter(outDim, inDim, init = Gaussian, initValueScale = wScale)
b = LearnableParameter(outDim, 1, init = fixedValue, value = bValue)
sc = LearnableParameter(outDim, 1, init = fixedValue, value = scValue)
m = LearnableParameter(outDim, 1, init = fixedValue, value = 0, learningRateMultiplier = 0)
isd = LearnableParameter(outDim, 1, init = fixedValue, value = 0, learningRateMultiplier = 0)
t = Times(W, x)
bn = BatchNormalization(t, sc, b, m, isd, eval = false, spatial = false, normalizationTimeConstant = bnTimeConst)
y = RectifiedLinear(bn)
]
# Fully-connected layer.
DnnLayer(inDim, outDim, x, parmScale) = [
W = LearnableParameter(outDim, inDim, init="uniform", initValueScale=parmScale)
b = LearnableParameter(outDim, 1, init="uniform", initValueScale=parmScale)
t = Times(W, x)
z = Plus(t, b)
]
]
DNN=[
# First Layer : DNN Layer with Sigmoid Activation (300 -> 300 neurons)
hidden_dim_1 = 300
hidden_1 = DnnSigmoidLayer(nFeatures, hidden_dim_1, featScaled, 1)
output_1 = DnnLayer(hidden_dim_1, hidden_dim_1, hidden_1, 1)
# Second Layer : DNN Layer With Sigmoid Activation (300 -> 300 neurons)
hidden_dim_2 = 300
hidden_2 = DnnSigmoidLayer(hidden_dim_1, hidden_dim_2, output_1, 1)
output_2 = DnnLayer(hidden_dim_1, hidden_dim_2, hidden_2, 1)
# Third Layer : DNN Layer With Sigmoid Activation (300 -> 7 neurons)
hidden_dim_3 = 300
hidden_3 = DnnSigmoidLayer(hidden_dim_2, hidden_dim_3, output_2, 1)
output_l = DnnLayer(hidden_dim_3, nLabels, hidden_3, 1)
ce = CrossEntropyWithSoftmax(labels, output_l)
err = ErrorPrediction(labels, output_l)
# Special Nodes
FeatureNodes = (features)
LabelNodes = (labels)
CriterionNodes = (ce)
EvalNodes = (err) OutputNodes = (output_l)
]
Le script ci-dessus définit une couche dont l'activation se fait via une sigmoïde (DNNSigmoidLayer) dont le résultat est stocké dans la variable hidden_1, et ce résultat est ensuite fourni à la couche de sortie (DNNLayer) qui prend en entrée hidden_dim_1 neurones, donc les valeurs sont issues de hidden_1. Le processus est identique pour la seconde et la troisième couche cachée. Enfin, la sortie output_l sera composée de nLabels
neurones alimentée par les 300 neurones de la troisième couche.
La macro CrossEntropyWithSoftmax permet de transcrire les valeurs de la couche de sortie en probabilités. Les valeurs sont alors comprises dans [0, 1], et la somme, égale à 1.
L'expression de l'enchainement des opérations est ici volontairement simple à titre d'exemple, mais vous pouvez y mettre toutes sortes d'opérations qui vous sont utiles et pertinente. ( Un exemple plus concret de définition utilisé pour le challenge MNIST (Modified National Institute of Standards and Technology) (Cf. précédent billet) est consultable ici. )
Sachez enfin que, par convention, ces fichiers de définition « avancés » portent l'extension .ndl.
Contrôle de l'apprentissage
Ce contrôle s'opère au travers de la macro SGD qui permet de définir comment seront mis à jour les différentes pondérations entre les neurones des différentes couches. ( Vous trouverez plus de détails ici. )
SGD = [
epochSize = 0
minibatchSize = 25
learningRatesPerMB = 0.5:0.2*20:0.1
momentumPerMB = 0.9
dropoutRate = 0.0
maxEpochs = 10
]
Les paramètres sont les suivants :
- epochSize : Ce paramètre précise le nombre d'exemples à sélectionner à chaque itération de l'algorithme (0 indique tous les exemples/lignes dans le jeu de données)
- maxEpochs : Ce paramètre indique le nombre d'itérations à opérer
- minibatchSize : Ce paramètre contrôle le nombre de mise à jour qui sera fait sur les pondérations du modèle. Plus il est important, plus le nombre de mise à jour sera important, et donc l'apprentissage long.
- dropoutRate : Ce paramètre permet de contrôler le sur-apprentissage ou sur-ajustement (overfitting). ( Vous trouverez plus d'informations ici. )
- learningRatePerMB : Il s'agit du pas d'apprentissage lors de la mise à jour des pondérations
- momentumPerMB : Ce paramètre précise le poids accordé à la précédente valeur lors de la mise à jour des pondérations
Gestion des entrées et sorties
Enfin, CNTK intègre au cœur de son moteur la gestion et la dé-sérialisation des données. Plusieurs formats sont supportés de base :
- UCIFastReader : Lecture de fichiers dont les colonnes sont séparées par un délimiteur.
- HTLMLFReader : Lecture de fichiers associés à la reconnaissance vocale, générés via le kit HTK (Hidden Markov Model Toolkit).
- LMReader : Lecture de fichiers textuels
- LUReader : Quasi identique que LMReader, à la différence près que l'entrée et la sortie diffèrent
- ImageReader : Lecture de fichiers images comme son nom l'indique
Toutes ces opérations de lectures sont regroupées au sein de la macro reader qui permet de définir le format, le fichier source, ainsi que les différents paramètres propres à chaque format.
Mise en œuvre de CNTK pour notre problématique
Comme indiqué en fin de la première partie du billet, nous sommes partis sur une topologie avec 3 couches cachées, comptant chacune 300 neurones. Les données d'entrée sont les spectrogrammes des différents axes sur une fenêtre de 10 secondes. Nous avons ainsi 774 variables et une dernière correspondant à l'activité.
L'apprentissage se fait simplement en invoquant CNTK.exe via une ligne de commande, en fournissant le fichier de configuration via le paramètre configFile.
CNTK.exe configFile=/path/to/config.cntk
L'exécution de l'apprentissage prend un temps non négligeable, environ 1 heure sur l'environnement d'exécution Microsoft Azure avec une machine virtuelle D3v2 sous Windows Server R2 2012.
( Comme indiqué ici, les instances de la série Dv2 sont basées sur la dernière génération de processeur Intel Xeon® E5-2673 v3 (Haswell) de 2,4 GHz et peuvent aller jusqu'à 3,2 GHz avec Intel Turbo Boost Technology 2.0. Les séries Dv2 sont idéales pour les applications nécessitant des processeurs plus rapides, de meilleures performances des disques locaux ou des mémoires plus volumineuses. )
La console vous permet alors de suivre l'évolution des différentes étapes du processus d'apprentissage :
Set Max Temp Mem Size For Convolution Nodes to 0 samples.
Starting Epoch 1: learning rate per sample = 0.003125 effective momentum = 0.900000 momentum as time constant = 303.7 samples
starting epoch 0 at record count 0, and file position 0
already there from last epoch
Starting minibatch loop.
RandomOrdering: 58 retries for 384 elements (15.1%) to ensure window condition
RandomOrdering: recached sequence for seed 0: 65, 163, ...
Epoch[ 1 of 1000]-Minibatch[ 1- 10]: SamplesSeen = 320; TrainLossPerSample = 2.03669014; EvalErr[0]PerSample = 0.75625000; TotalTime = 0.0525s; SamplesPerSecond = 6098.7
Finished Epoch[ 1 of 1000]: [Training Set] TrainLossPerSample = 1.99511; EvalErrPerSample = 0.75; AvgLearningRatePerSample = 0.003125; EpochTime=0.064001
Starting Epoch 2: learning rate per sample = 0.003125 effective momentum = 0.900000 momentum as time constant = 303.7 samples
starting epoch 1 at record count 384, and file position 24
already there from last epoch
Starting minibatch loop.
RandomOrdering: 73 retries for 384 elements (19.0%) to ensure window condition
RandomOrdering: recached sequence for seed 1: 139, 31, ...
Epoch[ 2 of 1000]-Minibatch[ 1- 10, 100.00%]: SamplesSeen = 320; TrainLossPerSample = 1.75856056; EvalErr[0]PerSample = 0.69375000; TotalTime = 0.0936s; SamplesPerSecond = 3417.9
Finished Epoch[ 2 of 1000]: [Training Set] TrainLossPerSample = 1.7688302; EvalErrPerSample = 0.70052087; AvgLearningRatePerSample = 0.003125; EpochTime=0.110412
Starting Epoch 3: learning rate per sample = 0.003125 effective momentum = 0.900000 momentum as time constant = 303.7 samples
starting epoch 2 at record count 768, and file position 48
already there from last epoch
Figure 2 : Sortie des différentes itérations de la macro "train"
La sortie précédente reprend certaines informations fournies dans votre fichier de configuration dans la macro train.
De plus, pour chaque itération de l'algorithme (Epoch), vous êtes en mesure de visualiser la convergence de l'apprentissage (via TrainLossPerSample), ainsi que des métadonnées sur la taille de l'échantillon utilisé pour cette itération, la vitesse de lecture des données, le Momentum utilisé pour la descente de gradient, etc.
Ces informations sont très utiles si vous utilisez des variables dynamiques pour contrôler l'apprentissage (taux d'apprentissage décroissant, Momentum dynamique, etc.)
Si vous avez ajouté une macro test, CNTK va alors procéder à l'évaluation du modèle :
Post-processing network complete.
evalNodeNames are not specified, using all the default evalnodes and training criterion nodes.
Allocating matrices for forward and/or backward propagation.
UCIFastReader: Starting at epoch 0, counting lines to determine record count...
360 records found.
starting epoch 0 at record count 0, and file position 0
already there from last epoch
Minibatch[1-1]: Samples Seen = 360 err: ErrorPrediction/Sample = 0.24444444 ce: CrossEntropyWithSoftmax/Sample = 0.75110236
Final Results: Minibatch[1-1]: Samples Seen = 360 err: ErrorPrediction/Sample = 0.24444444 ce: CrossEntropyWithSoftmax/Sample = 0.75110236 Perplexity = 2.119335
COMPLETED
Figure 3 : Sortie de la macro "test"
Parmi les informations affichées, l'erreur de prédiction moyenne ainsi que l'entropie croisée vous fourniront une représentation plus « haut niveau » de la performance de votre modèle.
La perplexité est une autre mesure de l'adéquation de votre modèle à la distribution de probabilité de vos données. (Dans ce cas une perplexité plus faible indique que le modèle est plus en adéquation avec la distribution de probabilité soumise.)
Enfin, la macro plot vous permet de générer une représentation graphique de l'enchainement des opérations dans votre réseau de neurones sous la forme d'un graphe DAG (Directed Acyclic Graph).
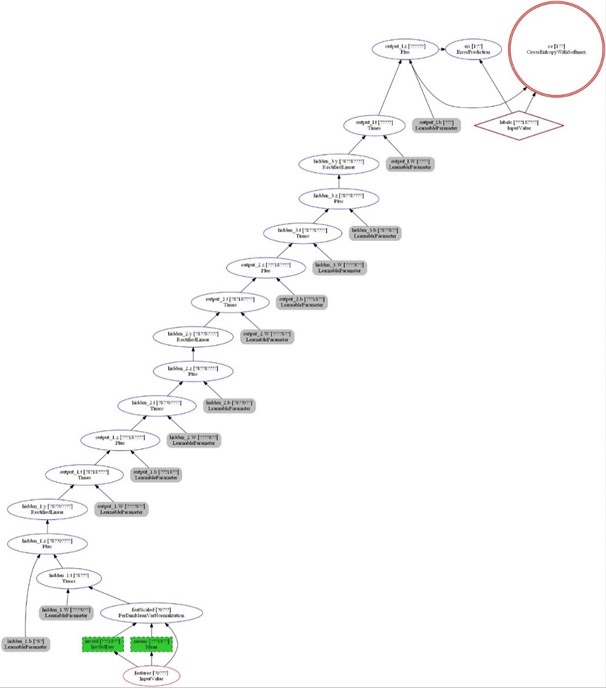
Figure 4 : DAG des opérations du réseau de neurones
Remarque : La génération d'un tel diagramme est soumise à la condition que les binaires de GraphViz soient disponibles sur votre environnement d'exécution. Une fois ces derniers installés, il vous faudra renseigner la macro plot de la manière suivante (Veillez à bien rajouter la macro dans le plan d'exécution de CNTK dans la macro command) :
Activity_Tracker_Plot = [
action = "plot"
outputFile="$OutputDir$/topologies/nn_topo.jpg"
outputDotFile="$OutputDir$/topologies/nn_topo.dot"
renderCmd="C:\Users\t-mofunt\Tools\GraphViz\bin\dot.exe -Tjpg -o"
]
La documentation complète de la commande plot est disponible ici.
Utilisation d'Azure pour un suivi en temps réel de l'activité
Nous évoquions ci-avant le type D3 de machine virtuelle au sein de la série Dv2dans l'environnement Microsoft Azure.
Pour poursuivre avec cet environnement, voyons à présent comment bénéficier de la puissance de Microsoft Azure afin d'utiliser CNTK pour suivre en temps réel l'activité de personnes.
L'idée est de ne plus reposer sur notre jeu de données de l'UCI mais de s'appuyer au contraire sur d'une hypothétique app mobile déployée sur les différents smartphones. Ceci permet de reboucler sur la solution initiale que nous nous proposions de mettre en œuvre de la première partie de ce billet.
Pour ce faire, une telle mise en œuvre supposerait de déployer les composants suivants proposés par l'environnement Microsoft Azure :
- Une machine virtuelle permettant d'héberger notre application intégrant le moteur de CNTK ainsi que le modèle – ce qui était implicitement suggéré jusqu'ici ;
- Un compte de stockage pour héberger les données transitoires issues de l'accéléromètre des différents smartphones utilisés ainsi que le résultat de la prédiction ;
- Un service bus de type Concentrateur d'évènements (Event Hub) permettant d'acheminer les données issues des différents accéléromètres vers l'environnement Microsoft Azure.
( Vous pouvez utiliser la machine virtuelle mise à disposition par Microsoft. Si vous n'êtes pas familier(e) avec cette dernière, nous vous invitons à lire ou à relire la série de billets qui lui est consacré sur ce même blog : Mise en place d'une machine virtuelle dédiée à l'analyse de données – 1ère partie, 2nde partie, et 3ième partie )
Sur cette base, il est dès lors possible d'envisager d'émettre les données des différents smartphones vers Microsoft Azure au travers du concentrateur d'évènements qui en assure l'ingestion.
CNTK exécuté depuis la machine virtuelle est consommateur de ce concentrateur et met en application le modèle sur les données présentées.
Enfin, une fois la prédiction effectuée, les données sont sauvegardées dans une base de données à des fins de reporting.
Le schéma global et le plan d'action associé pour aller plus loin serait le suivant :
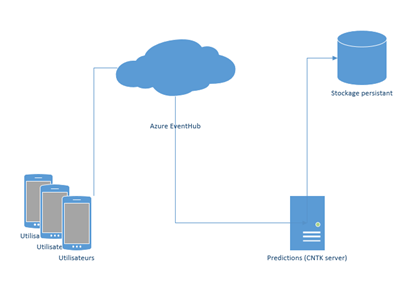
Figure 5 : Plan d'action
Nous nous en arrêtons cependant ici ; ces éléments allant au-delà de ce que nous souhaitions illustrer.
En guise de conclusion
Au travers de ce billet, et plus particulièrement de cette seconde partie, nous avons essayé de vous donner les éléments nécessaires pour prendre en main les différents outils de Deep Learning proposée par Microsoft et plus particulièrement CNTK. Comme vous avez pu le constater, CNTK propose une abstraction totale du modèle de calcul utilisé. Peu importe que vous utilisiez un CPU, un GPU, ou même une version distribuée de ces dernières, la configuration reste la même.
Le projet CNTK est encore récent et en amélioration constante. Parmi les récents ajouts, nous pouvons souligner l'ajout d'un wrapper Python au projet. Ce dernier est encore à l'état expérimental, mais d'ores et déjà utilisable, via le repo/la forge communautaire Github.
A ce jour, le projet CNTK est utilisé au sein de Microsoft sur des projets concrets nécessitant l'utilisation d'un cluster de calcul sur GPU au travers d'Azure GPU Lab. Il est à noter, que les applications sur GPU dans Azure seront bientôt proposée au grand public, comme en témoigne cette vidéo d'introduction ici.
Aussi, vous trouverez une documentation complète et en constante évolution sur le repo/la forge communautaire Github officiel du projet CNTK.